Применение искусственного интеллекта для улучшения диагностики и лечения воспалительных заболеваний кишечника
Nasim Sadat Seyed Tabib, Matthew Madgwick, Padhmanand Sudhakar, Bram Verstockt, Tamas Korcsmaros, Séverine Vermeire
Перевод из журнала BMJ Gut (оригинал).
Аннотация
Воспалительные заболевания кишечника (ВЗК) остаютсязагадкой для современной медицины. Сложность их этиопатогенеза обеспечивается сочетанием множества факторов, включая генетические особенности человека, изменения кишечного микробиома и влияние окружающей среды.
Не менее проблематичной является терапия ВЗК, несмотря на большие успехи медицины в этом направлении.
Достижения науки и современные технологии стали катализатором для улучшения медицинской помощи пациентам сВЗК. Появлениеискусственного интеллекта, в частности, машинного обучения, позволяет по-новому взглянуть на диагностику, лечение и прогнозирование исходов указанныхзаболеваний, с учетом необходимости индивидуального подхода в ведении каждого пациента.
Настоящий обзор посвящен применению машинного обучения для решения различных задач в исследованиях, посвященных ВЗК. Проблемы, которые позволяет решить внедрение искусственного интеллекта, включают разделение пациентов на группы риска и стратификацию тяжести заболевания, прогнозирование его течения и ответа на терапию.
Это способствует улучшению качества жизни пациентов с ВЗК исозданию более безопасных методов лечения. В обзоре также выделены проблемы, связанные с машинным обучением в клинических исследованиях ВЗК.
"Большие данные" в изучении ВЗК
Персонализированная медицина основана на индивидуальном подходе к каждому пациенту. С этой целью проводится разделение пациентов на основетипов и степени тяжести заболевания, вероятности его прогрессирования и ответа на лечение. Основой такого разделения традиционно являются данные клинических, лабораторных иинструментальных исследований. 1,2
Под типамиВЗК подразумевают болезнь Крона (БК) и язвенный колит (ЯК). Особенности клинического течения и сложность морфологической диагностики этих заболеваний часто затрудняют своевременное лечение. Ввиду этого ученые во всем мире ищут способыулучшения диагностики и терапии ВЗК с помощью современных технологий. 3-6
Перспективным для решения указанных задач является применение информационных технологий в здравоохранении. Современные достижения науки позволяют обрабатывать значительные объемы информации, с помощью компьютера подбирая соответствующие методы, что в совокупности обозначается термином "большие данные"(bigdata).7
За последние десятилетия производство и доступность данныхв системе здравоохранения заметно в��зросло, главным образом, из-за технологических достижений и снижения затрат на получение данных.
В отношении ВЗК наиболее важными источниками "больших данных"служат когортные исследования, клинические испытания, электронные медицинские карты. Используются также базы данных по метаболическому профилированию пациентов, включая геномику, транскриптомику, протеомику и метаболомику.7,8
Применение "больших данных" при ВЗК позволяет исследователям выявлять связанные с болезнью тенденции и ассоциации, строить прогностические модели, что важно для развития персонализированной медицины. Однако, ввиду сложности технологии, интерпретация полученных данных не является тривиальной. Требуются подходы, которые могут обнаружить скрытые закономерности в больших и сложных наборах данных.8
Классические статистические методы характеризуются недостаточной эффективностьюв объяснении причин ВЗК и предрасполагающих к болезни факторов. Для решения данной проблемы ученые применяют новые аналитические методы. 9 Как правило, они разделяются на две основные группы, а именно: системная биология и машинное обучение. Это более мощные, но при этом и более гибкие подходы в науке о биомедицинских данных.10,11
Системная биология сосредоточена вокруг математического моделирования, объясняющего целостность сложной биологической системы. Этот метод помогает в идентификации патофизиологических механизмов ВЗК. 12 Он подразумевает использование молекулярных сетей для выявления связей между экспрессией генов, образованием белков и метаболитов.13
Например, с применением метода системной биологии ученые обнаружили кластеры генов, повышающихриск ЯК. Они проанализировали связь между взаимодействиями, характерными для заболевания белков, иэкспрессией генов, повышающихриск ЯК, объединив их в одну сеть. 14
Применение искусственного интеллекта – самый прогрессивный на сегодня подход среди информационных технологий в здравоохранении. Машинное обучение представляет собой область искусственного интеллекта, с помощью которого происходит построение алгоритмов, способных обучаться и на основании этого выявлять закономерности и принимать соответствующие решения.11,15
В последнее десятилетие машинное обучение привлекало внимание ученых, занимающихся исследованиями ВЗК, благодаря возможности изучать сложные модели и делать прогнозы. Было предпринято множество попыток использования наборов клинических и метаболических данных для улучшения понимания патофизиологии ВЗК. 16 Новейшие разработки привели к интеграции "больших данных" и молекулярных сетей с использованием машинного обучения.
Преимуществом машинного обучения служит использование автоматических функций для анализа данных, в то время как подходы системной биологии должны программироваться вручную. Кроме того, с помощью машинного обучения можно интегрировать несколько предикторов для идентификации подмножества исходных данных.11-13
Алгоритмы машинного обучения в медицине позволяют исследовать большие объемы данных и классифицировать пациентов на основе выявляемых различий. С применением указанного метода может производиться одновременный анализ генома, транскриптома, протеома, эпигенома, иммунома и микробиомапациента(Рисунок 1).
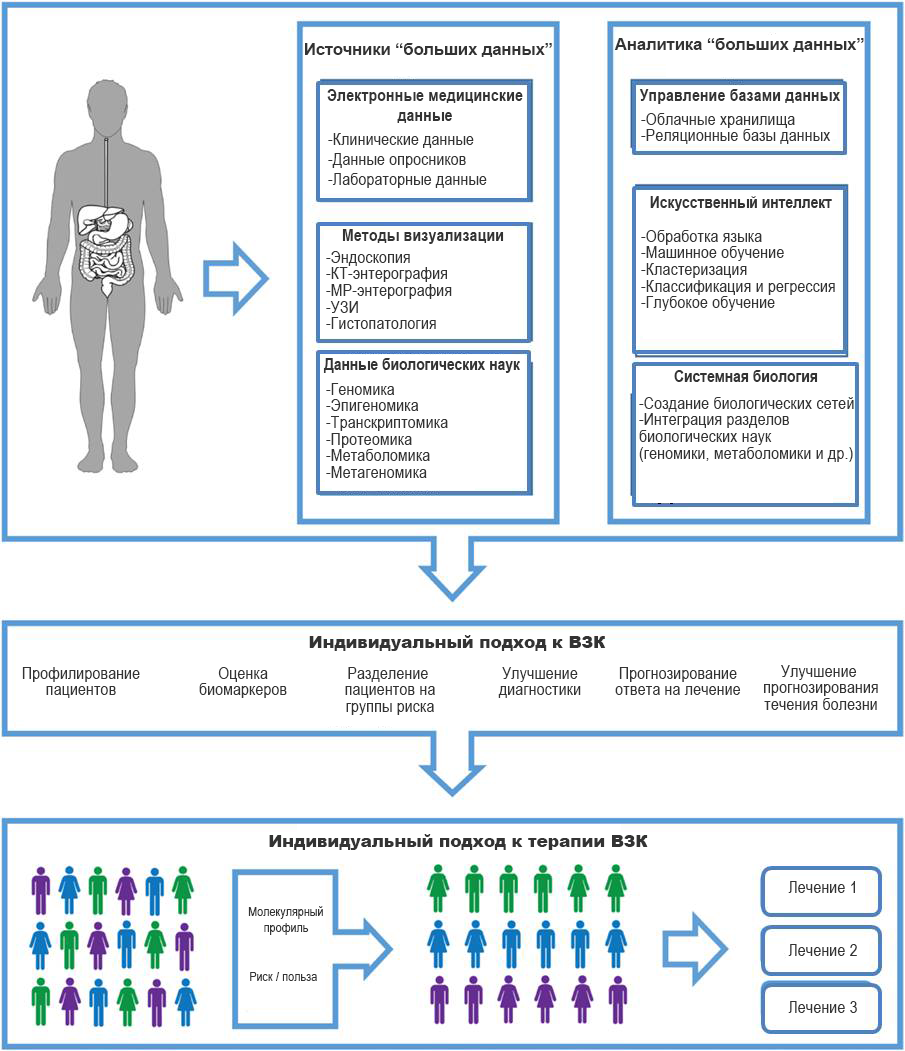
Рисунок 1. Роль машинного обучения в формировании индивидуального подхода к обследованию и лечению пациентов с ВЗК. Машинное обучение и системная биология способствуют развитию персонализированного подхода в лечении ВЗК с помощью разделения пациентов на группы по тяжести болезни и прогнозирования их ответа на лечение
Роль искусственного интеллекта в изучении биологических путей развития ВЗК
На сегодняшний день поиск генетических детерминант ВЗК продолжается, в том числе с применением системной биологии и машинного обучения. 16-19
Основной проблемой исследований, в которых проверяется взаимосвязь генов с развитием болезни, является дисбаланс между количеством анализируемых образцов и количеством однонуклеотидных полиморфизмов (SNP). Кроме того, классическая связь генотип-фенотип при высокой статистической достоверности упускает факт генетической изменчивости.
Тем не менее, машинное обучение позволяет с высокой достоверностью обнаружить генетические маркеры заболеваний. 19-22 Такое исследование было проведено с использованием данных проекта Immunochip Международного сообщества по изучению генетики ВЗК. Авторы построили модели, предсказывающие риск ВЗК на основании 573 однонуклеотидных полиморфизмов SNP, обнаруженных при БК и 366 однонуклеотидных полиморфизмов при ЯК.23
Еще одно исследование, проведенное Обществом по изучению генетики ВЗК Великобритании,на 8000 человек (4280 пациентов и 3652 человек в контрольной группе) применило метод опорных векторов для поиска новых генетических вариантов ВЗК. В результате обнаружена миссенс-мутация гена ADCY7, связанная с ЯК. 24
Искусственный интеллект может быть также использован для оценки влияния геномных мутаций на транскрипцию и образование белков (транскриптом и протеом), что вносит вклад в раскрытие тайн патогенеза ВЗК .25-31
Транскриптомный анализ образцов подвздошной кишки и толстой кишки человека позволил лучше понять роль различных метаболических путей, способствующих развитию воспаления при ВЗК. 32-42 С помощью анализа сети межбелковых взаимодействий было доказано, что гены по-разному экспрессируются в воспаленной и невоспаленной ткани кишечника. 32-33
Yuan et al сообщили об обнаружении 41 гена, связанного с ВЗК. Данные были получены с применением методов системной биологии и машинного обучения. В поиске новых генетических маркеров ВЗК ученые применили многоступенчатый анализ. В первую очередь, они распределили гены в соответствии их связи с диагнозом и с другими генами. Затем, применив классификатор машинного обучения, исследователи выделили набор из 21 гена, дающих высокую прогностическую точность развития ВЗК.41
Кроме того, с помощью протеомики, то есть изучения сетей межбелковых взаимодействий, в исследовании было определено функциональное сходство протеинов, закодированных в генетическом материале. Это позволило выявить еще 20 генов, потенциально вносящих вклад в развитие ВЗК.41
Применение машинного обучения для изучениякишечного микробиома при ВЗК
Микробиота кишечника, в состав которой входят кишечные бактерии, грибы,археи и вирусы, является важной частью желудочно-кишечного тракта и играет существенную роль в поддержании здоровья человека. 43,44
Изменение состава микроорганизмов, известное как дисбиоз, служит одним из важных признаков ВЗК. Характерной чертой нарушения составакишечной микробиоты при ВЗК служит сокращение ее видового разнообразия. Среди бактерий в пораженном кишечнике преобладает тип Proteobacteria,к которому относят, например, кишечную палочку. Отмечается снижение численности бактерий типа Firmicutes, которые вырабатывают противовоспалительные короткоцепочечные жирные кислоты. 44-47
Гораздо меньше известно в настоящее время о влиянии вирусов на дисбиозу пациентов с ВЗК. 46 Последние достижения в технологии секвенирования и методах анализа данных позволили провести углубленный анализ микробиотысреди указанной группы пациентов. Для этого также использованы данные метагеномики, метатранскриптомики, метапротеомики и метаболомики.47-49
Zuo et al применили машинное обучение на основе кластеризации для определения вирусных метасообществ слизистой оболочки прямой кишки, полученных от пациентов с ЯК. В результате отмечено снижение разнообразия и численности вирусов, особенно среди видов Caudovirales. Тем не менее, два вида Caudovirales (Escherichiaphage и Enterobacteriaphage) чаще встречались среди пациентов с ЯК по сравнению со здоровыми людьми. Ученые объясняют это нарушением взаимодействия между вирусами и бактериями, что определяет одну из причин развития дисбиоза. 46
Количество исследований, связанных с применением метапротеомики и метаболомики, растет с каждым годом.Для улучшения точности анализа данных здесь также может применяться машинное обучение. Согласно полученным данным, оно улучшает точность кластерного анализа данных, применяемого для таксономической характеристики микробного сообщества. 44-49
Ученые стремятся интегрировать данные геномики, транскриптомики, метаболомики, сведения о композиции микробиома кишечника для построения более полной картины патогенеза ВЗК. 50 Например, Lloyd-Priceet al выполнили исследование особенностей состава и функциональной активности микрофлоры кишечника при ВЗК с применением двух аналитических подходов: системной биологии и машинного обучения.
В исследовании приняло участие 132 пациента, наблюдение за которыми осуществлялось в течение года. Проводилось молекулярное профилирование микробиоты участников. Авторы выявили характерное изменение соотношения между факультативными аэробами, число которых оказалось больше, и облигатными анаэробами.
Наряду с этим отмеченынарушения на молекулярном уровне, включая микробную транскрипцию (в пределах кластера клостридий) и изменение обмена желчных и короткоцепочечных жирных кислот. Дополнительно, ученые сообщили о заметных изменениях в составе ифункциях микробиоты в зависимости от активности заболевания.51
Современная парадигма ведения пациентов с ВЗК
Улучшение понимания патофизиологии ВЗК привело к созданию новых эффективных препаратов для их лечения. Исследуются новые молекулы, нацеленные на различные мишени, например, антицитокиновые и хемокиновые агенты. Лечение стволовыми клетками и с помощью трансплантации фекальной микробиоты также перестает быть областью строго научного приложения.
Индивидуальный подход к терапии ВЗК в зависимости от особенностей организма, течения заболевания и других параметров, становится нормой клинической практики. 52
Ведение пациентов с ВЗК включает три основных этапа: подтверждение диагноза, назначение лечения и контроль активности заболевания.
Наблюдение за активностью ВЗК в настоящий момент осуществляется с помощью оценки различных маркеров, таких какфекальный кальпротектин, С-реактивный белок, а также колоноскопия и другие технологии медицинской визуализации. 53,54
До сих пор решение о выборе терапевтической стратегии зависело от ответа на лечение и его переносимости. В свете последних инновационных разработок в терапии ВЗК стало возможно более точно подбирать подход к лечению и скринингу данных заболеваний.54,55
С этой целью может быть использовано машинное обучение. 55 Оно позволяет дать персональный прогноз развития заболевания и подобрать оптимальную терапевтическую стратегию. Согласно полученным данным, применение машинного обучения позволяет одновременно и улучшить медицинскую помощь, и уменьшить расход на ее оказание 56 (Рисунок 2).
Рисунок 2. Ведение пациентов с ВЗК от подтверждения диагноза и далее в течение жизни.Каждый этап ведения пациентов с ВЗК потенциально может быть улучшен с помощью машинного обучения за счет снижения социально-экономической нагрузки на пациентов, врачей и систему здравоохранения
Интересным примером является исследование Bremeretal, которые применили машинное обучение для прогнозирования индивидуального результата лечения ирасходовсреди пациентов с депрессивными расстройствами. Результаты продемонстрировали возможность применения компьютерных технологий для решения указанных задач 56.
В другом исследовании Wanget al на основе машинного обучения была разработана новая стратегия анализа мочи, которая значительно увеличила скорость обнаружения возбудителя трихомонады и позволила сэкономить не только время, но и расходы.57
Диагностика и стратификация риска ВЗК
Текущая парадигма классификации ВЗК, которая опирается на инвазивные исследования, в первую очередь,илеоколоноскопию с забором биопсии, не полностью охватываетширокий спектр проявленийэтих заболеваний ивариантов манифестации у конкретного пациента.
Новые исследования сосредоточены на выявлении и оценке потенциала неинвазивных диагностических маркеров для диагностики ВЗК, улучшения их классификации и дифференциальной диагностики. 56-59
В перспективе совмещение данных о молекулярных и клеточных сигналах с генетическим профилем человека с помощью машинного обучения может улучшить определение степени тяжести ВЗК.
Не менее интересным направлением служит определение набора генетических и других признаков, позволяющих отличить БК от ЯК.60
Montero-Meléndezetalуспешно применили машинное обучение для определения транскрипции генов, характерных для БК или ЯК, в биоптатах толстой кишки. 61
Другим примером служит использование модели машинного обучения ProbabilisticPathwayScore, основанный на выявлении генных взаимодействий и молекулярных путей, участвующих в развитии заболевания. Обнаружение совпадений и различий между ними служит еще одним ключом к развитию индивидуального подхода в диагностике ВЗК. 62
Протеомика также является перспективнымнаправлением в исследованиях по выявлению биомаркеров ВЗК. 63-66 Seeley et al изучили белковые сигнатуры тканей толстой кишки с применением классификатора машинного обучения, обученного на результатах масс-спектрометрии и данных морфологического исследования биоптатов. В результате была построена модель, позволяющая проводить дифференциальную диагностику между ЯК и БК с точностью 76,9%.63
МикроРНК являются частицами, блокирующими образование определенных белков за счет контроля экспрессии генов. Они обнаруживаются в каждом органе, ткани и жидкостях организма. Проведенные ранее исследования доказали, что при ВЗК функция многих микроРНК нарушена. Анализ микроРНК с помощью методик машинного обучения также служит потенциальным способом улучшения диагностики и стратификации ВЗК. 65,66
Plevy et al.объединили в своем исследовании генетические, серологические и воспалительные маркеры для создания диагностической модели, позволяющей отличить пациентов с ВЗК от здоровых людей. На основании данных анализа 1520 человек, с помощью машинного обучения авторы отобрали 17 статистически значимыхмаркеров ВЗК. 67
Аналогичные подходы могут применяться и для оценки влияниякишечного микробиома на течение ВЗК. Учеными разработаны программы на основании машинного обучения, такие как Многомерная ассоциация с линейными моделями (MaAsLin) и Метагеномный подход к прогнозированию, которые позволяют вносить клинические данные пациента, сведения о таксономической и филогенетической структуре микробиома. 68-70
В качестве примера, Gevers et al применили MaAsLin для создания модели, на основании которой можно дифференцировать изменения микробиома детей с БК и другими заболеваниями кишечника.71
Несмотря на то, что результаты выявления биомаркеров для улучшения диагностики ВЗК с применением искусственного интеллекта являются многообещающими, до того, как они смогут найти применение в клинической практике, еще далеко. Причиной этому служит множество сопутствующих факторов, в том числе неоднородность болезней, объединенных термином "воспалительные заболевания кишечника", сопутствующие заболевания, и, что не менее важно, отсутствие валидации результатов. 72-84
Достижения в технологиях визуализации
Распознавание изображений является одной из основных точек приложения искусственного интеллекта и имеет большие перспективы в области медицинской визуализации.
Метод так называемого глубокого обучения(deep learning) является наиболее удобным для подобного анализа ввиду его способности декодировать содержимоеизображений.
Его появление предопределилораспространение исследований с попыткойавтоматизировать интерпретацию и оценку данных, полученных при эндоскопии, морфологическом исследовании,компьютерной (КТ) и магнитно-резонансной томографии (МРТ). 85
Эндоскопическая картина воспаления, характеристика поражений и оценка заживления слизистой оболочки кишечника имеет важное значение для правильного ведения пациентов с ВЗК. Проблема состоит в субъективности восприятия визуальных данных. Оценка изображения компьютером могла бы способствовать большей объективности интерпретации исследований.
Такие исследования проведены в отношении ВЗК. Stidham et al применили глубокое обучение для эндоскопической оценки тяжести ЯК. Классификация данных осуществлялась с разделением на 2 группы: ремиссия (оценка по шкале Мейо 0 или 1) и обострение (оценка по шкале Мейо 2 или 3). В результате созданная модель показала диагностическую способность в определении тяжести ЯК, сопоставимую с заключениями опытных эндоскопистов. 86
Bossuyt et al использовали глубокое обучение для измерения степени покраснения путем выделения интенсивности и распределения красного цвета в различных слоях изображения, что позволило создать новую компьютерную систему оценки активности ЯК (Рисунок 3). 87
Рисунок 3. Применение искусственного интеллекта в медицинской визуализации. Графическое представление сегментации изображений с помощью глубокого обучения для определения границы участков воспаления в кишечнике.Верхняя часть рисунка демонстрирует эндоскопическое изображение болезни Крона толстой кишки, проявляющейся в виде изъязвлений и деформации слизистой оболочки кишки по типу «булыжной мостовой». Использование глубокого обучения на основе метода сегментации изображения воспаленных участков помогает обнаружить: изменения кишки по типу «булыжной мостовой» – серый цвет, изъязвления – красный цвет. Нижняя часть рисунка иллюстрирует гистопатологическое изображение стеноза воспаленной подвздошной кишки. Глубокое обучение может применяться для сегментации изображения и прогнозирования границ воспаленных областей: острая инфильтрация (язва) –красный цвет, утолщение мышечного слоя – синий цвет, гиперплазия адипоцитов – желтый цвет
Машинное обучение может также улучшить морфологическую диагностику ВЗК, что особенно важно для пациентов с неклассифицированным вариантом заболевания. 88
Аналогичная ситуация наблюдается в радиологии. Описание результатов КТ и МРТ при ВЗК достаточно субъективны; следовательно, оценка изображений компьютером могла бы улучшить точность диагностики.
Полуавтоматическое программное обеспечение для анализа изображений показало производительность, аналогичную опытным радиологам для оценки структурного повреждения кишечника при КТ-энтерографии брюшной полости у пациентов с БК. 89
Схожиеметоды и алгоритмы машинного обучения были применены для прогнозирования степени тяжести БК по данным МРТ брюшной полости. 90,91 Наряду с этим, машинное обучение можетпомочь с трудоемкой оценкой данных беспроводной капсульной эндоскопии. Это открывает путь для автоматизированного анализа капсульных эндоскопических изображений и выявления изменений, характерных для БК через обнаружение предопределенных структурных и текстурных изменений.92,93
В итоге, искусственный интеллект служит многообещающим методом в улучшении визуальной диагностики ВЗК и в будущем, вероятно, будет широко применяться в этой области клинической практики. 94
Прогнозирование течения ВЗК
Прогнозированиетечения заболевания имеет решающее значение для определения тактики ведения пациентов с ВЗК.
Возможность извлечения информации из электронных медицинских карт (ЭМК), такой как клиническая картина болезни, лабораторные анализы, результаты эндоскопического исследования, создает благоприятную среду для применения машинного обучения. 95-99
Исследование Reddyetal продемонстрировало возможность использования машинного обучения с включением данных из ЭМК для предсказания тяжести течения БК. 95 Согласно Waljee et al, похожая модель может использоваться для прогнозирования риска госпитализации пациентов с ВЗК. Ученые установили, что пожилой возраст, высокий уровень тромбоцитов и альбумина сыворотки крови, применение стероидов и госпитализация в анамнезе служат прогностически неблагоприятными признаками. 96
С помощью метода полногеномных ассоциаций и машинного обучения удалось определить четыре гена (локуса), достоверно связанных с неблагоприятным прогнозом БК, а именно FOXO3,XACT, IGFBP1 и MHC.100
Одно из исследований было посвящено изучению функции микроорганизмов из биоптатов кишечника у 20 детей, не получавших лечения. В эксперименте приняло участие также 20 пациентов из контрольной группы. Ученые обратили внимание на прогностическую ценность профилирования микробиома с использованием 16РНК-секвенирования, одновременно с этим метагеномный анализ позволил идентифицировать маркеры хорошего ответа на лечение. 101
Объединение данных геномики, метаболомики, сведений о составе микрофлоры кишечника при разных состояниях у пациентов с ВЗК обеспечивает многообещающую перспективу в области применения машинного обучения для прогнозирования течения ВЗК. 102,103 Увеличение объема данных и усовершенствование аналитических методов позволит прогнозировать течение ВЗК с большей точностью.
Прогнозирование ответа на терапию ВЗК
В последние десятилетия были предприняты огромные усилия, чтобы предсказать ответ на терапию ВЗК и подобрать оптимальное лечение.
В настоящее время оценка клинической эффективности и риска побочных эффектов тиопуринов, препаратов с узким терапевтическим окном, проводится с помощью анализа крови и мониторинга уровня метаболита 6-метилмеркаптопурина.
Интеграция клинических и лабораторных данныхс помощью машинного обучения позволила ученым создать модель для выявления среди пациентов тех, кто ответит или не ответит на терапию тиопуринами. Кроме того, разработанный алгоритм привел к снижениючастоты госпитализаций по поводу ВЗК и уменьшению дозы стероидов.104, 105
Используя данные клинических исследований GEMINI I и GEMINI II по применению ведолизумаба у пациентов с ВЗК, Waljee et al разработалимодель машинного обучения, включившую демографические, клинические и лабораторные данные для прогнозирования вероятности достижения эндоскопической ремиссии без кортикостероидов у пациентов с ЯК и БК. В результате, наиболее значимыми прогностическими маркерами у пациентов с ЯК оказались низкие уровни фекального кальпротектина иальбумина, а средипациентов с БК– низкий уровень сывороточного С-реактивного белка и альбумина.106, 107
Аналогично предыдущему исследованию, Zarringhalam et al применили машинное обучение для выделения прогностических биомаркеров ответа на терапию инфликсимабом при рефрактерном к другому лечению ЯК. С помощью алгоритмов машинного обучения авторы определили, что гамма-интерферон, липополисахарид(ЛПС) и фактор некроза опухоли-альфа (ФНО-альфа) служат ключевыми регуляторами активации генов, ответственных за развитие ВЗК. 108 Отсутствие ответа на терапию у пациентов с ВЗК было связано с повышенной экспрессией генов, регулирующихся ФНО-альфа, а также активацией клеток иммунной системы из-за повышенной активности ЛПС, продуцируемого кишечной микрофлорой.109,110
Таким образом, исследования с применением алгоритмов машинного обучения для прогнозирования ответа на терапию у пациентов с ВЗК также стремятся к объединению данных геномики, транскриптомики, метаболомикии сведений о композиции кишечной микрофлоры для максимальной полноты и точности результатов. 111,112
Выводы
Искусственный интеллект предоставляет большие возможности для развития индивидуального подхода в медицине. Этому способствует накопление "больших данных"области геномики, транскриптомики, метаболомики и микробиологии. Прилагаются огромные усилия, чтобы сделать машинное обучение доступным способом обработки клинических ибиомедицинских данных в исследованиях. 113-118
Благодаря применению искусственного интеллектаученые уже получили возможность одновременно разрабатывать новые методы лечения, принимать обоснованные решения о назначении той или иной терапии и определять степени тяжести заболеваний.
Тем не менее, имеются проблемы в применении искусственного интеллекта в медицине. Несмотря на то, что существующие "большие данные" позволили интегрировать важную информацию из разных областей науки, впереди есть еще большой путь для внедрения машинного обучения в рутинную клиническую практику.
Это особенно актуально, учитывая тот факт, что современные подходы к диагностике и лечению болезней основываются на персонализированном подходе.
Появление "больших данных" и способов их анализа привело к возникновению множества гипотез и исследований. Однако многие из них требуют подтверждения результатов на когортах больших размеров. В будущем возможно создание биологических банков для хранения различных типов данных, включая генетические, молекулярные, клинические и серологические. Это требует объединения усилий ученых на национальном и международном уровне.
Таким образом, основой для развития машинного обучения для улучшения диагностики и лечения ВЗК служит координация между исследователями и финансирующими учреждениями. Сотрудничество в этой области обеспечит созданиеразносторонних наборов данных из образцов одних и тех же людей. Согласованный сбор, хранение и использование метаданных пациентов и медицинских карт также является важнейшей задачей.
Обеспечивая интеграцию данных, машинное обучение и системная биология предоставляют уникальныевозможности для изучения течения ВЗК.
Литература
1 Korcsmaros T, Schneider MV, Superti-Furga G. Next generation of network medicine:interdisciplinary signaling approaches. Integr Biol 2017;9:97–108.
2 Weersma RK, Xavier RJ, Vermeire S, et al. Multiomics analyses to deliver the most effective treatment to every patient with inflammatory bowel disease.Gastroenterology 2018;155:e1–4.
3 Ananthakrishnan AN. Epidemiology and risk factors for IBD. Nat Rev Gastroenterol Hepatol 2015;12:205–17.
4 de Souza HSP, Fiocchi C. Immunopathogenesis of IBD: current state of the art. NatRev Gastroenterol Hepatol 2016;13:13–27.
5 Ananthakrishnan AN, Bernstein CN, Iliopoulos D, et al. Environmental triggers in IBD:a review of progress and evidence. Nat Rev Gastroenterol Hepatol 2018;15:39–49.
6 Gecse KB, Vermeire S. Differential diagnosis of inflammatory bowel disease:imitations and complications. Lancet Gastroenterol Hepatol 2018;3:644–53.
7 Raghupathi W, Raghupathi V. Big data analytics in healthcare: promise and potential.Health Inf Sci Syst 2014;2:3.
8 Brooks J, Watson A, Korcsmaros T. Omics approaches to identify potential biomarkers of inflammatory diseases in the focal adhesion complex. Genomics Proteomics Bioinformatics 2017;15:101–9.
9 Gligorijevi? V, Pr?ulj N. Methods for biological data integration: perspectives and challenges. J R Soc Interface 2015;12:20150571.
10 Ideker T, Galitski T, Hood L. A new approach to decoding life: systems biology. Annu Rev Genomics Hum Genet 2001;2:343–72.
11 Camacho DM, Collins KM, Powers RK, et al. Next-Generation machine learning for biological networks. Cell 2018;173:1581–92.
12 Hood L, Tian Q. Systems approaches to biology and disease enable translational systems medicine. Genomics Proteomics Bioinformatics 2012;10:181–5.
13 Ngiam KY, Khor IW. Big data and machine learning algorithms for health-care delivery. Lancet Oncol 2019;20:e262–73.
14 Brooks J, Modos D, Sudhakar P, et al. A systems genomics approach to uncover patient-specific pathogenic pathways and proteins in a complex disease. bioRxiv 2019;692269.
15 Sorzano COS, Vargas J, Pascual-Montano AD. A survey of dimensionality reduction techniques. ArXiv 2014:abs/1403.2.
16 Tedjo DI, Smolinska A, Savelkoul PH, et al. The fecal microbiota as a biomarker for disease activity in Crohn’s disease. Sci Rep 2016;6:35216.
17 Mirkov MU, Verstockt B, Cleynen I. Genetics of inflammatory bowel disease: beyond NOD2. Lancet Gastroenterol Hepatol 2017;2:224–34.
18 Ogura Y, Bonen DK, Inohara N, et al. A frameshift mutation in NOD2 associated with susceptibility to Crohn’s disease. Nature 2001;411:603–6.
19 Shah SC, Kusiak A. Data mining and genetic algorithm based gene/SNP selection. Artif Intell Med 2004;31:183–96.
20 Phuong TM, Lin Z, Altman RB. Choosing SNPs using feature selection. In: 2005 IEEE Computational Systems Bioinformatics Conference (CSB’05). IEEE 2005:301–9.
21 Long N, Gianola D, Rosa GJM, et al. Machine learning classification procedure for selecting SNPs in genomic selection: application to early mortality in broilers. J AnimBreed Genet 2007;124:377–89.
22 Bermingham ML, Pong-Wong R, Spiliopoulou A, et al. Application of high-dimensionalfeature selection: evaluation for genomic prediction in man. Sci Rep2015;5:10312.
23 Wei Z, Wang W, Bradfield J, et al. Large sample size, wide variant spectrum, andadvanced machine-learning technique boost risk prediction for inflammatory boweldisease. Am J Hum Genet 2013;92:1008–12.
24 Luo Y, de Lange KM, Jostins L, et al. Exploring the genetic architecture ofinflammatory bowel disease by whole-genome sequencing identifies association at ADCY7. Nat Genet 2017;49:186–92.
25 Romagnoni A, Jégou S, Van Steen K, et al. Comparative performances of machinelearning methods for classifying Crohn disease patients using genome-wide genotyping data. Sci Rep 2019;9:10351.
26 Schwerd T, Bryant RV, Pandey S, et al. Nox1 loss-of- function genetic variants in patients with inflammatory bowel disease. Mucosal Immunol 2018;11:562–74.
27 Ghandi M, Lee D, Mohammad-NooriM, et al. Enhanced regulatory sequence prediction using gapped k-mer features. PLoS Comput Biol 2014;10:e1003711.
28 Alipanahi B, Delong A, Weirauch MT, et al. Predicting the sequence specificities of DNA- and RNA-binding proteins by deep learning. Nat Biotechnol 2015;33:831–8.
29 Zhou J, Troyanskaya OG. Predicting effects of noncoding variants with deep learning–based sequence model. Nat Methods 2015;12:931–4.
30 Kelley DR, Snoek J, Rinn JL. Basset: learning the regulatory code of the accessible genome with deep convolutional neural networks. Genome Res 2016;26:990–9.
31 Zou J, Huss M, Abid A, et al. A primer on deep learning in genomics. Nat Genet 2019;51:12–18.
32 Li XL, Zhou CY, Sun Y, et al. Bioinformatic analysis of potential candidates for therapy of inflammatory bowel disease. Eur Rev Med Pharmacol Sci 2015;19:4275–84.
34 Zhang B, Horvath S. A general framework for weighted gene co-expression network analysis. Stat Appl Genet Mol Biol 2005;4:Article17.
35 Langfelder P, Horvath S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics 2008;9:559.
36 Lin X, Li J, Zhao Q, et al. WGCNA reveals key roles of IL8 and MMP-9 in progression of involvement area in colon of patients with ulcerative colitis. Curr Med Sci2018;38:252–8.
37 Verstockt S, De Hertogh G, Van der Goten J, et al. Gene and Mirna Regulatory Networks During Different Stages of Crohn’s Disease. J Crohn’s Colitis 2019;13:916–30.
38 Verstockt B, Verstockt S, Creyns B, et al. Mucosal IL13RA2 expression predicts nonresponse to anti-TNF therapy in Crohn’s disease. Aliment Pharmacol Ther 2019;49:572–81.
39 Thiele I, Swainston N, Fleming RMT, et al. A community-driven global reconstruction of human metabolism. Nat Biotechnol 2013;31:419–25.
40 Knecht C, Fretter C, Rosenstiel P, et al. Distinct metabolic network states manifest in the gene expression profiles of pediatric inflammatory bowel disease patients and controls. Sci Rep 2016;6:32584.
41 Yuan F, Zhang Y-H, Kong X-Y,et al. Identification of candidate genes related to inflammatory bowel disease using minimum redundancy maximum relevance, incremental feature selection, and the shortest-path approach. Biomed Res Int 2017;2017:1–15.
42 Isakov O, Dotan I, Ben-Shachar S. Machine Learning-Based gene prioritization identifies novel candidate risk genes for inflammatory bowel disease. Inflamm Bowel Dis 2017;23:1516–23.
43 Manichanh C, Borruel N, Casellas F, et al. The gut microbiota in IBD. Nat Rev Gastroenterol Hepatol 2012;9:599–608.
44 Halfvarson J, Brislawn CJ, Lamendella R, et al. Dynamics of the human gut microbiome in inflammatory bowel disease. Nat Microbiol 2017;2:17004.
45 Norman JM, Handley SA, Baldridge MT, et al. Disease-Specific alterations in the enteric Virome in inflammatory bowel disease. Cell 2015;160:447–60.
46 Zuo T, Lu X-J, Zhang Y, et al. Gut mucosal virome alterations in ulcerative colitis. Gut 2019;68:1169–79.
47 Greenblum S, Turnbaugh PJ, Borenstein E. Metagenomic systems biology of the human gut microbiome reveals topological shifts associated with obesity and inflammatory bowel disease. Proc Natl Acad Sci U S A 2012;109:594–9.
48 Li X, LeBlanc J, Elashoff D, et al. Microgeographic proteomic networks of the human colonic mucosa and their association with inflammatory bowel disease. Cell Mol Gastroenterol Hepatol 2016;2:567–83.
49 Erickson AR, Cantarel BL, Lamendella R, et al. Integrated Metagenomics/ Metaproteomics reveals human Host-Microbiota signatures of Crohn’s disease. PLoSOne 2012;7:e49138.
50 Häsler R, Sheibani-Tezerji R, Sinha A, et al. Uncoupling of mucosal gene regulation, mRNA splicing and adherent microbiota signatures in inflammatory bowel disease. Gut 2017;66:2087–97.
51 Lloyd-Price J, Arze C, Ananthakrishnan AN, et al. Multi-Omics of the gut microbial ecosystem in inflammatory bowel diseases. Nature 2019;569:655–62.
52 Verstockt B, Ferrante M, Vermeire S, et al. New treatment options for inflammatory bowel diseases. J Gastroenterol 2018;53:585–90.
53 Maaser C, Sturm A, Vavricka SR, et al. ECCO-ESGAR guideline for diagnostic assessment in IBD Part 1: initial diagnosis, monitoring of known IBD, detection of complications. J Crohn’s Colitis 2019;13:144–64.
54 Sturm A, Maaser C, Calabrese E, et al. ECCO-ESGAR guideline for diagnostic assessment in IBD Part 2: IBD scores and general principles and technical aspects. J Crohn’s Colitis 2019;13:273–84.
55 Colombel J-F, Panaccione R, Bossuyt P, et al. Effect of tight control management on Crohn’s disease (calm): a multicentre, randomised, controlled phase 3 trial. Lancet 2017;390:2779–89.
56 Bremer V, Becker D, Kolovos S, et al. Predicting therapy success and costs for personalized treatment recommendations using baseline characteristics: data-driven analysis. J Med Internet Res 2018;20:e10275.
57 Wang H-Y, Hung C-C, Chen C-H, et al. Increase Trichomonas vaginalis detection based on urine routine analysis through a machine learning approach. Sci Rep 2019;9.
58 Cleynen I, Boucher G, Jostins L, et al. Inherited determinants of Crohn’s disease and ulcerative colitis phenotypes: a genetic association study. Lancet 2016;387:156–67.
59 Jeong C-S, Kim D. Inferring Crohn’s disease association from exome sequences by integrating biological knowledge. BMC Med Genomics 2016;9:35.
60 Medina I, Montaner D, Tarraga J, et al. Prophet, a web-based tool for class prediction using microarray data. Bioinformatics 2007;23:390–1.
61 Montero-Meléndez T, Llor X, García-Planella E, et al. Identification of novel predictor classifiers for inflammatory bowel disease by gene expression profiling. PLoS One 2013;8:e76235.
62 Han L, Maciejewski M, Brockel C, et al. A probabilistic pathway score (PROPS) for classification with applications to inflammatory bowel disease. Bioinformatics 2018;34:985–93.
63 Seeley EH, Washington MK, Caprioli RM, et al. Proteomic patterns of colonic mucosal tissues delineate Crohn’s colitis and ulcerative colitis. Proteomics Clin Appl 2013;7:541–9.
64 Cao B, Zhou X, Ma J, et al. Role of miRNAs in inflammatory bowel disease. Dig Dis Sci 2017;62:1426–38.
65 Duttagupta R, DiRienzo S, Jiang R, et al. Genome-Wide maps of circulating miRNA biomarkers for ulcerative colitis. PLoS One 2012;7:e31241.
66 Hübenthal M, Hemmrich-Stanisak G, Degenhardt F, et al. Sparse modeling reveals miRNA signatures for diagnostics of inflammatory bowel disease. PLoS One 2015;10:e0140155.
67 Plevy S, Silverberg MS, Lockton S, et al. Combined serological, genetic, and inflammatory markers differentiate Non-IBD, Crohn?s disease, and ulcerative colitis patients. Inflamm Bowel Dis 2013;19:1139–48.
68 Morgan XC, Tickle TL, Sokol H, et al. Dysfunction of the intestinal microbiome in inflammatory bowel disease and treatment. Genome Biol 2012;13:R79.
69 Pasolli E, Truong DT, Malik F, et al. Machine learning meta-analysis of large metagenomic datasets: tools and biological insights. PLoS Comput Biol 2016;12:e1004977.
70 Fioravanti D, Giarratano Y, Maggio V, et al. Phylogenetic convolutional neuralnetworks in metagenomics. BMC Bioinformatics 2018;19:49.
71 Gevers D, Kugathasan S, Denson LA, et al. The treatment-naive microbiome in new-onset Crohn’s disease. Cell Host Microbe 2014;15:382–92.
72 | Pfizer for Professionals. PROSIT-BIO. Available: https://www. pfizerpro. co. uk/ product/ inflectra/ ulcerative- colitis/ support/ prosit- bio-0 [Accessed 23 Aug 2019].
73 1000 IBD. Available: https:// 1000ibd. org/ [Accessed 23 Aug 2019].
74 Spekhorst LM, Imhann F, Festen EAM, et al. Cohort profile: design and first results of the Dutch IBD Biobank: a prospective, nationwide Biobank of patients with inflammatory bowel disease. BMJ Open 2017;7:e016695.
75 Chaparro M, Ramas M, Benítez JM, et al. Extracolonic cancer in inflammatory bowel disease: data from the GETECCU Eneida registry. Am J Gastroenterol 2017;112:1135–43.
76 Beaulieu DB, Ananthakrishnan AN, Martin C, et al. Use of biologic therapy by pregnant women with inflammatory bowel disease does not affect infant response to vaccines. Clin Gastroenterol Hepatol 2018;16:99–105.
77 IBDMDB. Home IBDMDB. Available: https:// ibdmdb. org/ [Accessed 23 Aug 2019].
78 PREdiCCt. Home. Available: https://www. predicct. co. uk/ [Accessed 23 Aug 2019].
79 CSIBD PRISM registry Hospital, Boston, MA. Available: https://www. massgeneral. org/ csibd/ cores/ clinical. aspx [Accessed 9 Jan 2020].
80 IBD BioResource. Translating today’s science into tomorrow’s treatments. Available:https://www. ibdbioresource. nihr. ac. uk/ [Accessed 9 Jan 2020].
81 Home - SWISS IBDcohort. Available: http://www. ibdcohort. ch/ [Accessed 9 Jan 2020].
82 Swibreg. Patient. Available: http://www. swibreg. se/ [Accessed 9 Jan 2020].
83 EPIMAD : le plus grand registre au monde – Observatoire National des MICI. Available: http://www. observatoire- crohn- rch. fr/ epimad- le- plus- grand- registre- demalades-au- monde/ [Accessed 9 Jan 2020].
84 Study management - Competence Network for Bowel Diseases. Available: http://www. kompetenznetz-darmerkrankungen. de/ Studienmanagement [Accessed 9 Jan 2020].
85 Bossuyt P, Vermeire S, Bisschops R. Scoring endoscopic disease activity in IBD: artificial intelligence sees more and better than we do. Gut 2019:gutjnl-2019-318235.
86 Stidham RW, Liu W, Bishu S, et al. Performance of a deep learning model vs human reviewers in grading endoscopic disease severity of patients with ulcerative colitis. JAMA Netw Open 2019;2:e193963.
87 Bossuyt P, Nakase H, Vermeire S, et al. 436 - Automated Digital Calculation of Endoscopic Inflammation in Ulcerative Colitis: Results of the Red Density Study. Gastroenterology 2018;154:S98–9.
88 Bossuyt P, Nakase H, Vermeire S, et al. Automatic, computer-aided determination of endoscopic and histological inflammation in patients with mild to moderate ulcerative colitis based on red density. Gut 2020:gutjnl-2019-320056.
89 Stidham RW, Enchakalody B, Waljee AK, et al. Assessing Small Bowel Stricturing and Morphology in Crohn’s Disease Using Semi-automated Image Analysis. Inflamm Bowel Dis;11.
90 Tielbeek JAW, Vos FM, Stoker J. A computer-assisted model for detection of MRI signs of Crohn’s disease activity: future or fiction? Abdom Imaging 2012;37:967–73.
91 Mahapatra D, Schüffler PJ, Tielbeek JAW, et al. Semi-supervised and active learning for automatic segmentation of Crohn’s disease. Med Image Comput Comput Assist Interv 2013;16:214–21.
92 Kumar R, Qian Zhao Q, Seshamani S, et al. Assessment of Crohn’s Disease Lesions in Wireless Capsule Endoscopy Images. IEEE Trans Biomed Eng 2012;59:355–62.
93 Charisis VS, Hadjileontiadis LJ. Potential of hybrid adaptive filtering in inflammatory lesion detection from capsule endoscopy images. WJG 2016;22:8641.
94 Bielecki C, Bocklitz TW, Schmitt M, et al. Classification of inflammatory bowel diseases by means of Raman spectroscopic imaging of epithelium cells. J Biomed Opt 2012;17:0760301.
95 Reddy BK, Delen D, Agrawal RK. Predicting and explaining inflammation in Crohn’s disease patients using predictive analytics methods and electronic medical record data. Health Informatics J 2019;25:1201–18.
96 Waljee AK, Lipson R, Wiitala WL, et al. Predicting hospitalization and outpatient corticosteroid use in inflammatory bowel disease patients using machine learning. Inflamm Bowel Dis 2018;24:45–53.
97 Ananthakrishnan AN, Cai T, Savova G, et al. Improving case definition of Crohn?s disease and ulcerative colitis in electronic medical records using natural language processing. Inflamm Bowel Dis 2013;19:1411–20.
98 Cai T, Lin T-C, Bond A, et al. The association between arthralgia and vedolizumab using natural language processing. Inflamm Bowel Dis 2018;24:2242–6.
99 Hou JK, Chang M, Nguyen T, et al. Automated identification of surveillance colonoscopy in inflammatory bowel disease using natural language processing. Dig Dis Sci 2013;58:936–41.
100 Lee JC, Biasci D, Roberts R, et al. Genome-Wide association study identifies distinct genetic contributions to prognosis and susceptibility in Crohn’s disease. Nat Genet 2017;49:262–8.
101 Tang MS, Bowcutt R, Leung JM, et al. Integrated analysis of biopsies from inflammatory bowel disease patients identifies SAA1 as a link between mucosal microbes with Th17 and Th22 cells. Inflamm Bowel Dis 2017;23:1544–54.
102 Douglas GM, Hansen R, Jones CMA, et al. Multi-omics differentially classify disease state and treatment outcome in pediatric Crohn’s disease. Microbiome 2018;6:13
103 Cushing KC, Mclean R, McDonald KG, et al. Predicting Risk of Postoperative Disease Recurrence in Crohn’s Disease: Patients With Indolent Crohn’s Disease Have Distinct Whole Transcriptome Profiles at the Time of First Surgery. Inflamm Bowel Dis 2019;25:180–93.
104 Waljee AK, Joyce JC, Wang S, et al. Algorithms Outperform metabolite tests in predicting response of patients with inflammatory bowel disease to thiopurines. Clin Gastroenterol Hepatol 2010;8:143–50.
105 Waljee AK, Sauder K, Patel A, et al. Machine learning algorithms for objective remission and clinical outcomes with thiopurines. J Crohns Colitis 2017;11:801–10.
106 Waljee AK, Liu B, Sauder K, et al. Predicting corticosteroid-free endoscopic remission with vedolizumab in ulcerative colitis. Aliment Pharmacol Ther 2018;47:763–72.
107 Waljee AK, Liu B, Sauder K, et al. Predicting Corticosteroid-Free Biologic Remission with Vedolizumab in Crohn’s Disease. Inflamm Bowel Dis 2018;24:1185–92.
108 Zarringhalam K, Enayetallah A, Reddy P, et al. Robust clinical outcome prediction based on Bayesian analysis of transcriptional profiles and prior causal networks. Bioinformatics 2014;30:i69–77.
109 Vázquez-Baeza Y, Callewaert C, Debelius J, et al. Impacts of the human gut microbiome on therapeutics. Annu Rev Pharmacol Toxicol 2018;58:253–70.
110 Doherty MK, Ding T, Koumpouras C, et al. Fecal Microbiota Signatures Are Associated with Response to Ustekinumab Therapy among Crohn’s Disease Patients. MBio 2018;9:e02120–17.
111 Shaw KA, Bertha M, Hofmekler T, et al. Dysbiosis, inflammation, and response to treatment: a longitudinal study of pediatric subjects with newly diagnosed inflammatory bowel disease. Genome Med 2016;8:75.
112 Verstockt B, Sudahakar P, Creyns B, et al. DOP70 An integrated multi-omics biomarker predicting endoscopic response in ustekinumab treated patients with Crohn’s disease. J Crohn’s Colitis 2019.
113 Piening BD, Zhou W, Contrepois K, et al. Integrative personal omics profiles during periods of weight gain and loss. Cell Syst 2018;6:157–70.
114 Zhou W, Sailani MR, Contrepois K, et al. Longitudinal multi-omics of host-microbe dynamics in prediabetes. Nature 2019;569:663–71.
115 Allez M, Tieng V, Nakazawa A, et al. CD4+NKG2D+ T cells in Crohn’s disease mediate inflammatory and cytotoxic responses through MICA interactions. Gastroenterology 2007;132:2346–58.
116 Lee JC, Lyons PA, McKinney EF, et al. Gene expression profiling of CD8+ T cells predicts prognosis in patients with Crohn disease and ulcerative colitis. J Clin Invest 2011;121:4170–9.
117 Imam T, Park S, Kaplan MH, et al. Effector T helper cell subsets in inflammatory bowel diseases. Front Immunol 2018;9:1212.
118 Chapuy L, Bsat M, Rubio M, et al. IL-12 and mucosal CD14+ monocyte-like cells induce IL-8 in colonic memory CD4+ T cells of patients with Ulcerative colitis but not Crohn’s disease. J Crohn’s Colitis.
Купить номер с этой статьей в pdf